Nguyễn Văn Ba1, Phạm Thị Phương Mai1, Trần Thị Thu Thủy1, Phạm Thu Thảo1và Phạm Doãn Lân1*
1 Viện Chăn nuôi * Tác giả liên hệ: TS. Phạm Doãn Lân, Phó Viện trưởng Viện Chăn nuôi. Điện thoại: Email: pdlanvn@yahoo.com
Ngày nhận bài báo: 12/03/2021 - Ngày nhận bài phản biện: 22/03/2021
Ngày bài báo được chấp nhận đăng: 14/04/2021
TÓM TẮT
Việt Nam là quốc gia có nguồn gen vịt bản địa khá đa dạng và phong phú. Nghiên cứu được thực hiện nhằm phân tích tính đa dạng di truyền gen D-loop ty thể ở một số giống vịt nuôi tại Việt Nam, đồng thời phân tích mối quan hệ di truyền và phát sinh loài giữa vịt Việt Nam với một số giống vịt trên thế giới. Tổng số 276 mẫu máu của 15 giống vịt được thu thập và tách chiết ADN tổng số. Đoạn gen D-loop (481bp) ty thể được tiến hành giải trình tự trực tiếp. Kết quả nghiên cứu cho thấy: trong 15 giống vịt đã xác định được 44 haplotype với 31 điểm đa hình. Đa dạng di truyền cao được tìm thấy ở giống vịt trời Bắc Mỹ và vịt Minh Hương trong khi đó đa dạng di truyền thấp thể hiện ở giống vịt Cỏ và Huba. Cây phát sinh chủng loại dựa vào haplotype gen D-loop giữa vịt Việt Nam và vịt một số nước là bằng chứng phân tử cho thấy chúng đều có nguồn gốc từ vịt Anas platyrhynchos.
Từ khoá: Gen D-loop ty thể, haplotype, đa dạng di truyền.
ABSTRACT
Genetic diversity of Mitochondrial D-loop gene in some Vietnamese native duck breedsVietnam has a diverse range of indigenous duck genetic resources. The study was conducted to analyze the genetic diversity of Mitochondrial D-loop gene in some duck breeds and to analyze the phylogenetic relationships between Vietnamese native ducks and some duck breeds in the world. Total 276 blood samples from 15 duck breeds were collected and DNA was extracted. A fragment of Mitochondrial D-loop gene (481bp) was sequenced directly for 276 samples. The results showed that 44 different haplotypes of D-loop gene were identifiedbased on 31 polymorphisms. The highest haplotype diversity of D-loop gene was found in Bắc Mỹ and Minh Hương duck breed and the lowest haplotype diversity of D-loop gene was found in Co and Huba duck breed. The phylogenetic tree provided evidence that Vietnamese native duck breeds origin was from Anas platyrhynchos.
Keywords: Mitochondrial Dloop gene, duck, genetic diversity.
1. ĐẶT VẤN ĐỀ
Sử dụng các chỉ thị di truyền phân tử để đánh giá sự đa dạng di truyền giữa các giống và trong bản thân các giống vật nuôi ở mức độ phân tử sẽ giúp định hướng cho việc quản lý, bảo tồn và sử dụng nguồn gen động vật nuôi một cách hữu ích. ADN ty thể là một trong những chỉ thị di truyền được sử dụng rộng rãi trong nghiên cứu về đa dạng di truyền quần thể. Ở ADN ty thể, đặc biệt là ở một số đoạn của vùng điều khiển (D-loop), cytochrome b và locus 12S rARN chúng có mức độ tiến hóa rất nhanh và đã được chứng minh là rất hiệu quả trong việc nghiên cứu về cấu trúc và đa dạng di truyền. Phân tử ADN ty thể không trải qua bất kỳ một trạng thái tái tổ hợp nào.
Hơn nữa, hệ gen ty thể có đặc tính di truyền theo dòng mẹ ở hầu hết các loài và vì vậy mỗi một dòng gen ty thể có sự di truyền độc lập. Do đó, hệ gen ty thể rất phù hợp trong việc nghiên cứu về quan hệ nguồn gốc tiến hoá (Ouithavon, 2009). Việt Nam là một nước có mức độ đa dạng sinh học cao với nhiều loài đặc hữu. Nguồn gen vịt nội ở Việt Nam cũng khá nhiều giống như: Vịt Kỳ Lừa, Đốm, Bạch Tuyết, Cỏ, Sín Chéng, Minh Hương, Bầu Bến, Bầu Quỳ, Cổ Lũng, Mốc, Ô Môn và Hòa Lan. Tuy nhiên, đa dạng sinh học tại Việt Nam trong những năm gần đây đang phải đối mặt với những mối đe dọa vô cùng nghiêm trọng từ các hoạt động của con người như khai thác quá mức, phá hủy và làm xuống cấp môi trường tự nhiên, ô nhiễm môi trường, sinh vật ngoại lai và gần đây tác động của biến đổi khí hậu.
Một số giống vịt đã biến mất hoặc nguy cấp như vịt Bạch Tuyết, Ô Môn, Kỳ Lừa. Để đối phó với những mối đe dọa về đa dạng sinh học này, chúng ta cần có những công cụ hữu hiệu giúp cho những nhà khoa học và những nhà quản lý có thể kiểm soát tốt hơn những hoạt động gây ảnh hưởng tới các loài hiện đang nguy cấp. Một trong những công cụ đang được các nước trên thế giới phát triển là dựa trên các kỹ thuật sinh học phân tử nghiên cứu marker ADN đặc trưng cho các loài đang được quan tâm (Stolpovskiy và Zakharov, 2017). Vì vậy, nghiên cứu này được thực hiện để phân tích đa dạng di truyền ở một số giống vịt bản địa và vịt nhập nội Việt Nam dựa trên vùng gen D-loop ty thể nhằm đánh giá sự đa dạng di truyền và mối quan hệ di truyền dòng mẹ của một số giống vịt ở Việt Nam đồng thời xác định mối quan hệ họ hàng giữa vịt Việt Nam với một số giống vịt trên thế giới.
2. VẬT LIỆU VÀ PHƯƠNG PHÁP NGHIÊN CỨU
2.1. Đối tượng
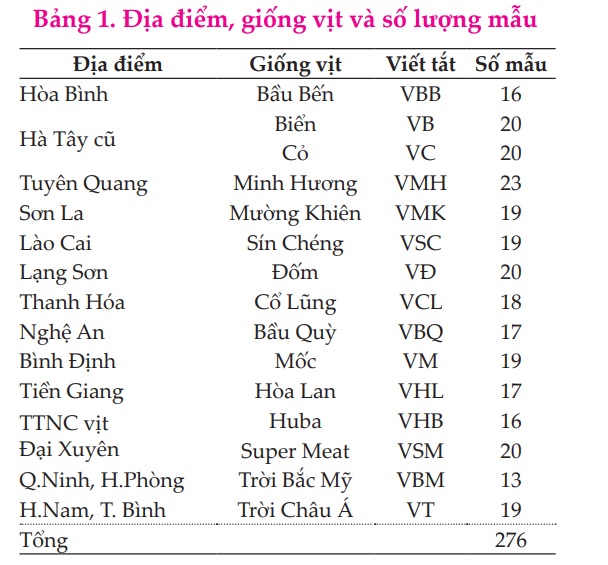
Thực hiện trên 15 giống vịt, được thu thập từ các vùng địa lý khác nhau trên lãnh thổ Việt Nam bao gồm: Hòa Bình, Hà Tây cũ, Hà Nam, Thái Bình, Tuyên Quang, Sơn La, Lào Cai, Lạng Sơn, Thanh Hóa, Nghệ An, Bình Định, Tiền Giang, Hải Phòng và Quảng Ninh (Bảng
1) với tổng cộng 276 mẫu máu được bảo quản trong dung dịch EDTA 0,5M để đảm bảo chất lượng mẫu trong quá trình vận chuyển. Sử dụng một số trình tự trên ngân hàng gen thế giới để phân tích so sánh mối quan hệ phát sinh chủng loài giữa vịt Việt Nam và thế giới cụ thể: 2 Haplotype vịt Thái Lan (EU013948, EU013952); 4 Haplotype vịt bản địa Trung Quốc (EF126702, EF126731, EU755252.1, KJ833586.1); 2 Haplotype vịt Indonesia (KX756168, KX712255); 2 Hapotype vịt Hàn Quốc (KU845272, KU845299); 1 Haplotype vịt Cộng Hòa Séc (JN811041); 1 Haplotype vịt Viễn Đông Nga (AY506947); 1 Haplotype Ngan (vịt Xiêm-EU755254)
2.2. Phương pháp
Tách chiết ADN: ADN tổng số được tách chiết từ mẫu máu bằng bộ kit DNeasy Blood & Tissue Kit từ Qiagen, được bảo quản ở nhiệt độ 2~8°C. Để kiểm tra nồng độ và độ tinh khiết của mẫu, tiến hành điện di ADN trên gel agarose 2% và máy quang phổ hấp thụ Nanodrop 2000.
Phản ứng PCR gen D-loop: Toàn bộ gen D-loop ty thể của 276 mẫu phân tích được khuếch đại bằng phản ứng PCR với cặp mồi xuôi ngược được sử dụng từ công bố của He và ctv (2008):5′- CCTACCTATCGGACTACCCTC -3′5′- GCAGGTGTGTCCAGGCTTAGA -3′
Sản phẩm của phản ứng PCR là một đoạn gen D-loop với trình tự dài 481bp. Phản ứng PCR được thực hiện với tổng thể tích 25µl: 2,5µl đệm PCR 10X; 2,5µl MgCl2; 2,5mM; 2,5µl dNTPs 2mM; 0,3µl Taq DNA Polymerase 1u/µl; 1µl mồi xuôi và 1µl mồi ngược 10pM, mẫu DNA 0,1µl 50-100ng. Phản ứng PCR được thực hiện theo chu trình nhiệt: 95oC trong 5 phút, 35 chu kỳ với 94oC 45 giây, 60oC 50 giây, 72oC 60 giây. Kết thúc phản ứng ở 72oC trong 10 phút.
2.4. Tinh sạch sản phẩm PCR và giải trình tự
Tinh sạch sản phẩm của phản ứng PCR được tiến hành bằng bộ kit tinh sạch của hãng Invitrogen. Quy trình được lập cho giải trình tự tự động theo module BigDye® Terminator™ sử dụng để làm sạch sau sequencing bằng kit Polymer: POP7 và Capillary loại 3130 & 3100 – Avent Capillary Array - 36cm trên hệ thống máy giải trình tự tự động AB 3130.
2.5. Phân tích kết quả
Dữ liệu thô của 276 mẫu phân tích gen D-loopty thể sau khi giải trình tự được xử lý bằng phần mềm BioEdit 6.0. Phần mềm DnaSP 5.10 được sử dụng để phân tích đa dạng các nucleotide và phát hiện các haplotype gen D-loop. Khoảng cách di truyền được tính theo phương pháp của Tamura và Nei (1993) và cây phát sinh chủng loại xây dựng bằng phương pháp NJ (Neighbor‒Joining) trong phần mềm MEGA 7.0.
3. KẾT QUẢ VÀ THẢO LUẬN
3.1. Đa dạng di truyền gen D-loop ty thể ở các giống vịt
Đoạn gen D-loop (481bp) sau khi giải trình tự được tiến hành xử lý loại bỏ đoạn đầu và đoạn cuối thu được trình tự đoạn gen D-loopdài 470bp của 15 giống vịt Việt Nam. Kết quả phân tích đa dạng di truyền trình tự gen D-loop ở các giống vịt được thể hiện ở Bảng 2.
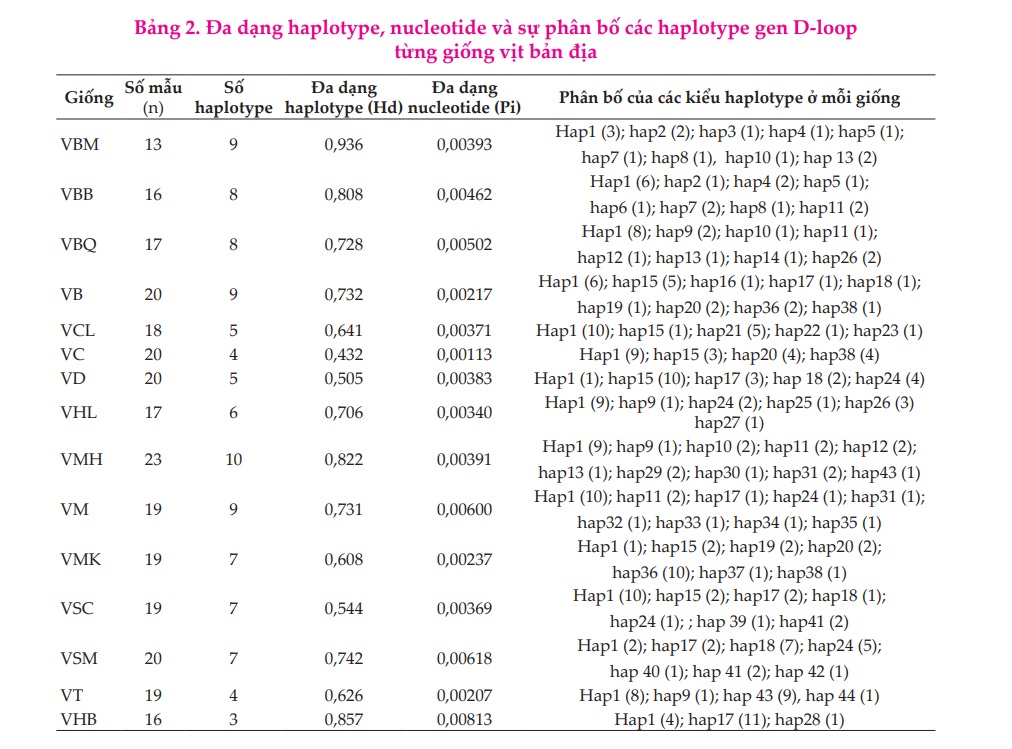
Trong 15 giống vịt được nghiên cứu, giống vịt Minh Hương (Tuyên Quang) có số haplotype nhiều nhất-phát hiện được 10 haplotype-với đa dạng haplotype Hd=0,822; tiếp đến là vịt Mốc và Trời Bắc Mỹ (cổ xanh) 9 haplotype; vịt Bầu Bến, Bầu Quỳ và Biển có 9 haplotype; vịt Mường Khiêng, Sín Chéng và Super Meat có 7 haplotype; vịt Hòa Lan có 6 haplotype; vịt Cổ Lũng và Đốm có 5 haplotype; vịt Cỏ và Trời
Châu Á (mỏ khuyết) có 4 haplotype; thấp nhất là vịt Huba chỉ có 3 haplotype. Giống vịt Minh Hương mặc dù có số haplotype nhiều nhất nhưng sự đa dạng haplotype lại thấp hơn so với vịt Trời Bắc Mỹ (Hd =0,936). Sự đa dạng nucleotide cao nhất ở giống vịt Super Meat (0,00618) và thấp nhất ở giống vịt Cỏ (0,00113).
Kết quả phân tích cho thấy các giống vịt bản địa Việt Nam có sự đa dạng cao hơn so với vịt ngoại và vịt trời. Vịt trời Bắc Mỹ có sự đa dạng cao hơn so với vịt trời Châu Á, điều này có thể lý giải do sự phổ biến của giống vịt trời Bắc Mỹ trên toàn thế giới hơn so với giống vịt trời châu Á. Trong 2 giống vịt ngoại thì vịt Super Meat có sự đa dạng cao hơn nhiều so với giống vịt Huba. Vịt Super Meat là giống vịt đã được nhập về Việt Nam từ lâu (những năm 90) còn giống vịt Huba có nguồn gốc từ Hungari và mới được nhập về Việt Nam từ năm 2019. Tổng hợp kết quả phân tích trình tự đoạn gen Dloop (470bp) ở 15 giống vịt cho thấy xuất hiện 44 haplotype (Bảng 2).
Qua bảng 2 cho thấy haplotype 1 là haplotype phổ biến nhất, xuất hiện ở tất cả các giống vịt 96/276 (chiếm 34,78%). Tiếp đến là haplotype 15 xuất hiện ở 6 giống vịt với số lượng là 23/276 (8,33%), haplotype 17 cũng xuất hiện ở 6 giống vịt với số lượng 20/276 (7,25%) và 19 hapotype chỉ xuất hiện ở 1 giống.
Từ dữ liệu phân tích trình tự gene D-loop, chúng tôi đã tiến hành xây dựng thành công cây phát sinh chủng loại giúp minh họa mối quan hệ di truyền của 15 giống vịt nghiên cứu (Hình 1).
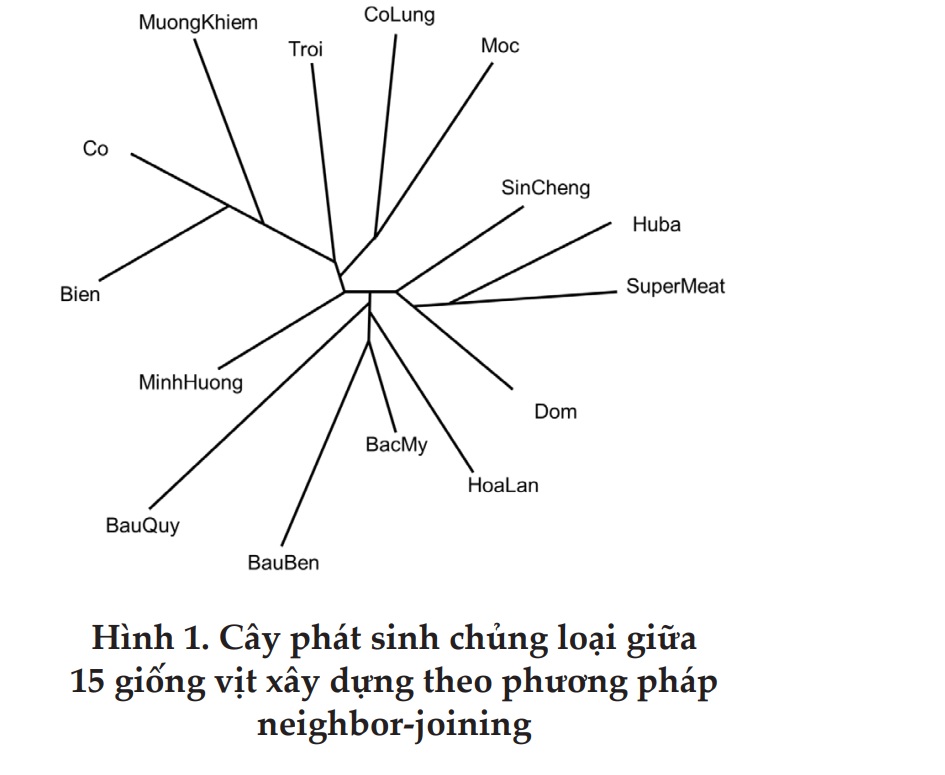
Cây phát sinh chủng loại theo dòng mẹ giữa 15 giống vịt nghiên cứu dựa trên trình tự đoạn gen Dloop được chia thành 2 nhóm chính cụ thể như sau:
- Nhóm 1 gồm 9 giống: Sín Chéng, Huba, Super Meat, Đốm, Hòa Lan, Trời Bắc Mỹ, Bầu Bến, Bầu Quỳ và Minh Hương.
- Nhóm 2 gồm 6 giống: Mường Khiêng, Trời Châu Á, Biển, Cỏ, Mốc và Cổ Lũng.
Qua hình 1 cho thấy trong 9 giống vịt ở nhóm 1 là những giống vịt “Cổ Xanh” tên thường gọi là vịt “Bầu” lại chia thành 2 nhóm nhỏ (nhóm phụ):
+ Nhóm phụ 1.1 gồm 4 giống: Super Meat, Huba, Sín Chéng và vịt Đốm;
+ Nhóm phụ 1.2 gồm 5 giống: Hòa Lan, Trời Bắc Mỹ, Bầu Bến, Bầu Quỳ và Minh Hương.
Trong 9 giống vịt “Bầu” trừ vịt Super Meat thì 8 giống còn lại đều có con trống khoang cổ xanh rất đặc trưng của dòng vịt “Bầu”. Mặc dù giống vịt Super Meat có màu lông toàn thân trắng tuyền, nhưng có hình dáng đặc trưng của vịt “Bầu” như chân ngắn, cổ ngắn, đầu to, dáng đuôi cong, bệt sệt. Hai giống vịt Đốm và Sín Chéng (phân bố ở biên giới phía Bắc giáp Trung Quốc) có quan hệ gần gũi vớinhóm vịt ngoại là Super Meat và Huba.
Sáu giống vịt nằm trong nhóm 2 phân bố thành 2 nhóm phụ:
+ Nhóm phụ 2.1 là những giống vịt thuộc nhóm vịt có tên thân thuộc là vịt “Cỏ” và có quan hệ gần gũi với vịt trời châu Á.
+ Nhóm phụ 2.2 là vịt ở miền Trung (vịt Mốc và vịt Cổ Lũng). Vịt Mốc phân bố ở Bình Định còn vịt Cổ Lũng phân bố ở Thanh Hóa.
3.2. Mối quan hệ di truyền giữa 15 giống vịt nghiên cứu với một số giống vịt trên thế giới.
Kết quả phân tích cho thấy mức độ đa dạng các haplotype ở các giống vịt bản địa Việt Nam khá cao, nghiên cứu đã xác định được tổng cộng 44 haplotype khác nhau trên 15 giống vịt phân tích. Trình tự 44 haplotype của 15 giống vịt trong nghiên cứu này được so sánh với 14 trình tự trên ngân hàng gen của một số giống vịt: Trung Quốc (EF126702, EF126731, EU755252.1 và KJ833586.1); Hàn Quốc (KU845272, KU845299); Indonesia (KX756168, KX712255); Thái Lan (EU013948, EU013952); Cộng Hòa Séc (JN811041), Viễn Đông Nga (AY506947) và Ngan (vịt Xiêm Cairina moschata EU755254).
Kết quả phân tích đã thu được 47 haplotype trong đó vịt Xiêm, vịt Viễn Đông Nga và vịt Cộng hòa Séc tạo thành 3 haplotype mới còn vịt Indonesia, Hàn Quốc, Trung Quốc, Thái Lan có trình tự trùng với haplotype 1 (haplotype phổ biến trong 44 haplotype) và haplotype 2 của vịt Việt Nam. Mối quan hệ di truyền giữa 47 haplotype được thể hiện ở
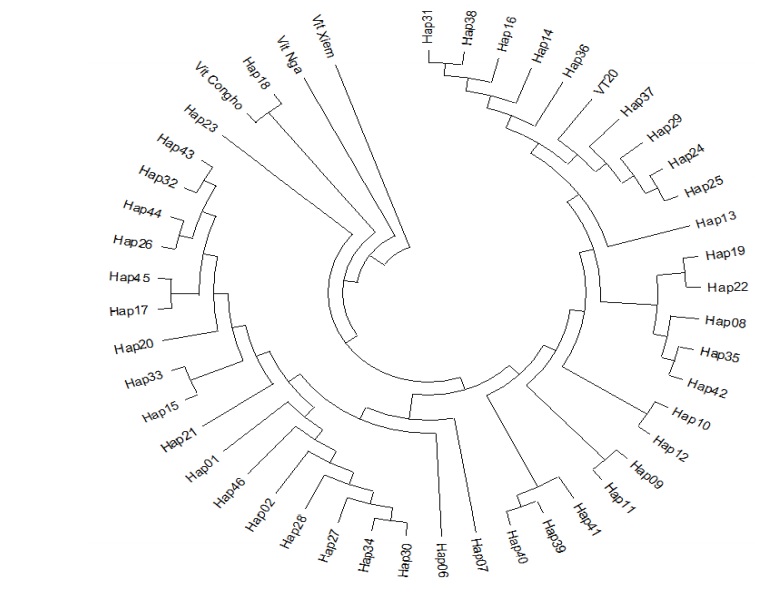
Theo Li và ctv (2006) cho rằng vịt nhà được thuần hóa từ vịt trời cổ xanh (wild mallard - Anas platyrhynchos). Sultana và ctv (2016) đã giải trình tự vùng Dloop ty thể để đánh giá đa dạng di truyền và phân tích mối quan hệ di truyền giữa các quần thể vịt Đông và Nam Á (Bangladesh, Trung Quốc, Hàn Quốc). Tất cả các quần thể này đều có quan hệ mật thiết với vịt trời (Anas platyrhynchos). Li và ctv (2010) đã phân tích vùng Dloop ty thể của 8 giống vịt bản địa Trung Quốc. Cây phát sinh loài NJ được xây dựng dựa trên 38 haplotypes (96 trình tự, bao gồm 12 trình tự của Anas Platyrhynchos, 6 trình tự của Anas zonorhyncha và 78 trình tự vịt bản địa Trung Quốc). Kết quả cho thấy nguồn gốc dòng mẹ của 8 giống vịt bản địa đều từ Anas platyrhynchos. Sultana và ctv (2016) đã giải trình tự vùng Dloop ty thể để đánh giá đa dạng di truyền và phân tích mối quan hệ di truyền giữa các quần thể vịt Đông và Nam Á (Bangladesh, Trung Quốc, Hàn Quốc). Tất cả các quần thể này đều có quan hệ mật thiết với vịt trời mallard (Anas platyrhynchos). Phân tích cây phả hệ theo phương pháp neighborjoining cho thấy nhữngquần thể vịt nội địa Đông Nam Á đã được thuần hóa từ vịt Anas platyrhynchos.
Kết quả phân tích của nghiên cứu này cho thấy bằng chứng phân tử về mối quan hệ di truyền theo dòng mẹ giữa 15 giống vịt Việt Nam với một số giống vịt thế giới: Vịt Việt Nam có mối quan hệ gần gũi với vịt Trung Quốc, Hàn Quốc, Indonessia, Thái Lan ( Anas platyrhynchos) và khác xa so với vịt mỏ khuyết vùng Viễn Đông Nga (Anas zonorhyncha) và vịt Xiêm (Cairina moschata).
4. KẾT LUẬN
Đã thành công trong việc khuếch đại và giải trình tự gen Dloop ty thể của 15 giống vịt là tiền đề cho việc nghiên cứu đa dạng di truyền và nguồn gốc của các giống vịt bản địa Việt Nam.
Mức độ đa dạng haplotype gen Dloop ở 15 giống vịt là khá cao: trong 15 giống phân tích đã xác định được 44 haplotype với 31 điểm đa hình. Đa dạng di truyền cao ở giống vịt trời Bắc Mỹ và vịt Minh Hương trong khi đó đa dạng di truyền thấp ở giống vịt Cỏ và vịt Huba.
Kết quả nghiên cứu là bằng chứng phân tử về mối quan hệ phát sinh chủng loại giữa vịt Việt Nam và vịt một số nước trên thế giới. Phân tích mối quan hệ phát sinh chủng loại qua so sánh 44 haplotype của 15 giống vịt cho thấy chúng đều có nguồn gốc từ vịt Anas platyrhynchos.
TÀI LIỆU THAM KHẢO
1. He Da-Qian, Zhu Qing, Shi-Yi Chen, Hui-Ying Wang, Yi-Ping Liu and Yong-Gang Yao (2008). A homogenous nature of native Chinese duck matrilineal pool. BMC Evolutionnary Biology, 8: 298. doi: 10.1186/1471-2148-8-298.
2. Li H.-F., Li B.C., Chen K.W., Yang N., Ma Y.H., Tang Q.P. and Tu Y.J. (2006). Study on Molecular Genetic Diversity of Native Duck Breeds in China. Acta Vet. Zoo. Sinica, 11: 1107-13.
3. Li H.-F., Zhu W.-Q., Song W.-T., Shu J.-T., Han W. and Chen K.-W. (2010). Molecular genetic diversity and origin of Chinese domestic duck breeds. Archives Ani. Bre., 53(5): 609-17.
4. Ouithavon K. (2009). Molecular phylogenetic relationships among Thai deer (subfamily Cervinae). A thesis for the degree of Doctor of Philosophy (Bioscience) Graduate School, Kasetsart University, Thailand.
5. Stolpovskiy Y.A. and Zakharov G.I.A. (2017). The problem of conservation of gene pools of domesticated animals. J. Gen. Bre., 21(4): 477-86.
6. Sultana H., Seo D.W., Bhuiyan M.S.A., Choi N.R., Hoque M.R., Heo K.N. and Lee J.H. (2016). Genetic diversity and phylogenetic analysis of South-East Asian duck populations based on the mtDNA D-loop sequences. Asian-Aus. J. Ani. Sci., 29(12): 1688.
7. Tamura K. and Nei M. (1993). Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol. Bio.Evo., 10(3): 512-26.