Trần Đức Hoàn1 *, Đoàn Thị Thảo1 , Nguyễn Thị Hương Giang1 và Nguyễn Đình Nguyên1
1 Trường Đại học Nông Lâm Bắc Giang * Tác giả liên hệ: TS. Trần Đức Hoàn, Khoa Chăn nuôi - Thú y, Trường Đại học Nông Lâm Bắc Giang; Điện thoại: 0965 679 819; Email: dr.hoan288@gmail.com
Ngày nhận bài báo: 30/03/2021 - Ngày nhận bài phản biện: 30/04/2021 Ngày bài báo được chấp nhận đăng: 04/05/2021
TÓM TẮT
Nghiên cứu được thực hiện nhằm ứng dụng kỹ thuật RT-PCR (Reverse Transcript-Polymerase Chain Reaction) để chẩn đoán bệnh dịch tả lợn cổ điển dựa trên đoạn gen ncE2 của virus. Giống virus được sử dụng trong nghiên cứu được phân lập từ lợn chết tại tỉnh Bắc Giang năm 2018. Cặp mồi đặc hiệu được thiết kế dựa trên trình tự nucleotide trên ngân hàng gen thế giới (NCBI).
Bằng phương pháp RT-PCR với các điều kiện tối ưu hóa, kết quả nghiên cứu cho thấy, virus dịch tả lợn cổ điển phát triển tốt trên môi trường tế bào PK15a trong DMEM bổ sung 5% huyết thanh bào thai bê. Giống virus được gây nhiễm trên môi trường tế bào theo hướng dẫn của tổ chức OIE. Phản ứng RT-PCR được thực hiện thành công với cặp mồi CSF324/326 ở nhiệt độ gắn mồi 50-60°C. Trình tự, gen ncE2 của virus dịch tả lợn được giải trình với độ dài 284bp, mã hóa 93 axít amin, độ tương đồng 99% so với chủng đã công bố trên ngân hàng gen NCBI. Kết quả nghiên cứu là cơ sở phân tích tính di truyền của virus dịch tả lợn cổ điển lưu hành tại Việt Nam.
Từ khóa: Gen ncE2, lợn, Reverse Transcript-Polymerase Chain Reaction, virus dịch tả.
ABSTRACT Detection of clasical swine fever virus base on ncE2 gene using Reverse Transcript[1]Polymerase Chain Reaction (RT-PCR) The study aimed to application of Reverse Transcript-Polymerase Chain Reaction technique to diagnose clasical swine fever disease base on E2 of virus gene fragment. The virus strain used in this study was isolated from dead pigs in Bac Giang province in 2018. A pair of specific pri[1]mers were designed basing on nucleotide sequences on NCBI (National Center for Biotechnology Information). Through out RT-PCR with the optimal condition of reaction, the results showed that, clasical swine fever virus could grow on the PK15a cell medium in DMEM supplement 5% calve embryo serum. Virus strain was infected on the cells medium according to Organization of International Epidemiology. RT-PCR reaction was successfully detected with the pair of primers CSF324/326 at the annealing temperature from 50-60°C. The seqencing of clasical swine fever virus ncE2 gene of this strain with 284bp, encoding 99 amino acids, similarity of 99% as compare with the published one in the Genebank. The results of the study is basic to analyze clasical swine fever virus genetic in Vietnam. Keywords: Gene ncE2, Reverse Transcript-Polymerase Chain Reaction, fever virus.
1. ĐẶT VẤN ĐỀ
Ngày nay, với những mô hình chăn nuôi quy mô công nghiệp ngày càng phát triển thì kéo theo đó dịch bệnh ngày càng trở nên phức tạp. Trong đó, bệnh dịch tả lợn cổ điển (Classical Swine Fever-CSF) là một trong những bệnh truyền nhiễm nguy hiểm trên lợn bởi sự lây lan nhanh, tỷ lệ chết cao (85- 100%) cho mọi lứa tuổi và gây ra những thiệt hại nặng nề về kinh tế (Nguyễn Thị Phương Duyên và ctv, 2001). Bệnh dịch tả lợn do virus thuộc chi Pestivirus, họ Flaviviridae gây ra; bệnh lây lan chủ yếu qua đường tiêu hóa và đường hô hấp do tiếp xúc trực tiếp với lợn ốm hoặc gián tiếp qua các chất bài tiết, thức ăn, nước uống, dụng cụ chăn nuôi (Nguyễn Tiến Dũng, 1999; Bhaskar và ctv, 2015). Ở Việt Nam, bệnh dịch tả lợn được phát hiện lần đầu tiên vào năm 1923-1924 bởi Houdener. Từ đó đến nay vẫn còn tồn tại phổ biến và luôn luôn là mối đe doạ nghiêm trọng đối với ngành chăn nuôi lợn nước ta (Đào Trọng Đạt và Nguyễn Tiến Dũng, 1985; Nguyễn Tiến Dũng, 1999).
Cho đến nay, công tác chẩn đoán, phòng và chống bệnh này vẫn là vấn đề nan giải. Trong công tác chẩn đoán hay phòng và trị bệnh, các phương pháp truyền thống dựa trên dịch tễ lâm sàng hiện ngày càng bộc lộ nhiều khuyết điểm và không thể theo kịp tình hình diễn biến của bệnh như hiện nay. Trong khi đó hiện nay việc áp dụng công nghệ sinh học vào chẩn đoán bệnh đang phát huy tác dụng rất hiệu quả đặc biệt là việc chẩn đoán nhanh và chính xác mầm bệnh và góp phần không nhỏ trong quá trình điều trị cũng như ngăn chặn dịch bệnh lấy lan.
Để phân tích chính xác virus gây bệnh dịch tả lợn với những virus khác cùng chi Pestivirus như BVDV, BDV..., kỹ thuật RTPCR trở nên hữu ích với những đoạn gen như 5’NTR, E2, NS5B trong việc phân biệt chủng virus dịch tả lợn và tổng kết sự phân biệt chủng virus dịch tả lợn ở nhiều nước trên thế giới kể cả châu Á, nghiên cứu này nhằm ứng dụng vào công tác chẩn đoán bệnh và tạo tiền đề cho những nghiên cứu xác định chủng, phân loại di truyền virus dịch tả lợn sau này.
2. VẬT LIỆU VÀ PHƯƠNG PHÁP NGHIÊN CỨU
2.1. Vật liệu
Virus dịch tả lợn, được phân lập từ lợn chết tại Bắc Giang năm 2018. Các dụng cụ và hóa chất phòng thí nghiệm cần thiết: Môi trường cho nuôi cấy tế bào; vật liệu, sinh phẩm dùng trong phản ứng RT-PCR và hóa chất dùng trong điện di ADN.
2.2. Phương pháp
2.2.1. Phương pháp nuôi cấy tế bào
Virus dịch tả lợn được nuôi cấy trên tế bào PK15a theo phương pháp nuôi cấy tế bào. Tế bào từ môi trường nitơ lỏng, được nuôi vào chai nuôi T25 bổ sung vừa đủ 6ml môi trường. Sau đó, nuôi trong tủ ấm 37°C 5% CO2 . Tế bào được theo dõi hàng ngày qua quan sát bằng kính hiển vi quang học. 2.2.2. Phương pháp gây nhiễm virus dịch tả lợn trên dòng tế bào PK15a Được thực hiện theo hướng dẫn của OIE gồm các bước như sau: Chuẩn bị tế bào PK15a bám đáy 100%. Gây nhiễm virus, nuôi trong tủ ấm 37°C 5% CO2 . Thu virus sau 4-5 ngày gây nhiễm.
2.2.3. Chiết tách, tinh sạch RNA của virus dịch tả lợn
Sử dụng kít thương mại chiết tách, tinh sạch RNA của virus dịch tả lợn làm “khuôn mẫu” cho việc tổng hợp cDNA, các bước thực hiện theo hướng dẫn của nhà sản xuất.
2.2.4. Phương pháp tổng hợp cDNA
Phản ứng tổng hợp cDNA từ khuôn mRNA được thực hiện như sau: Hỗn hợp phản ứng: Total RNA 5µl; 5mM dNTPs 2µl; H2 O 5µl. Đem ủ ở 65°C trong 5 phút, sau đó ủ trên đá 5 phút. Bổ sung 4µl 5X First-Strand Buffer; 1µl 0.1 M DTT; 1µl RNaseOUT; 1µl SuperScript III RT (200 U/µl). Trộn đều, rồi ủ ở 55°C trong 60 phút. Dừng phản ứng ở 70°C trong 15 phút, bổ sung 1µl (2U) của RNase H và ủ ở 37°C trong 20 phút.
2.2.5. Thiết kế mồi đặc hiệu gen vùng noncoding E2 của virus dịch tả lợn
Để thiết kế cặp mồi đặc hiệu gen noncoding E2 (ncE2) của virus dịch tả lợn, 20 trình tự gen E2 đã công bố trên ngân hàng dữ liệu NCBI.
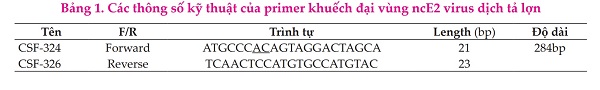
Trình tự ở 2 đầu 5’ và 3’ của gen đích được sử dụng để thiết kế mồi đặc hiệu cho vùng noncoding E2 virus dịch tả lợn. Trình tự của cặp mồi cho nhân gen vùng noncoding E2 virus dịch tả lợn với các thông số được thể hiện qua bảng 1.
2.2.6. Phương pháp PCR
Phương pháp PCR (sau sinh tổng hợp cDNA hoặc RT-PCR) được tiến hành trên cơ sở phương pháp đã được phổ biến truyền thống. Chu trình nhiệt như sau: Tiền khởi động 94°C trong 5 phút; duỗi mạch 94°C trong 30 giây; gắn mồi 60°C trong 30 giây; kéo dài 72°C trong 30 giây và kết thúc 72°C trong 10 phút. Sản phẩm phản ứng PCR được điện di trên gel Agarose 2% trong dung dịch TAE 1x.
2.2.7. Phương pháp giải trình tự gen
Sản phẩm PCR được tinh sạch theo kít được hướng dẫn của nhà sản xuất, rồi gửi tới Công ty Biotech, Hàn Quốc để giải trình tự gen của virus dịch tả lợn, được đọc trên phần mềm Chromas.
3. KẾT QUẢ VÀ THẢO LUẬN
3.1. Kết quả nuôi cấy tế bào
Để đảm bảo cho các thí nghiệm về gây nhiễm vi rút và nhân giống vi rút đạt kết quả tốt, một trong những bước quan trọng đầu tiên là phải có được tế bào nuôi ở trạng thái đang phát triển tốt và phủ đầy đáy chai 100% sau 1-2 ngày nuôi cấy. Vì vậy, lượng tế bào cấy chuyển ban đầu cần tối thiếu từ 2x105 tế bào/ ml đến 5x105 tế bào/ml, đồng thời trong quá trình nuôi cấy phải đảm bảo đầy đủ các điều kiện về dinh dưỡng, nhiệt độ, CO2 thích hợp.
Trong nghiên cứu này, tế bào PK15a được nuôi cấy trong môi trường DMEM bổ sung 5% huyết thanh bào thai bê phát triển khỏe mạnh và sau 2 ngày nuôi cấy tế bào PK15a đã bám đáy và mọc kín chai nuôi. Tế bào mạnh khỏe, phát triển tốt, hình thái đặc trưng là các hình đa giác, có rất ít tế bào chết. Hình thái quan sát được soi dưới kính hiển vi soi ngược cho thấy các tế bào này hoàn toàn khỏe mạnh, không bị tạp nhiễm và đủ tiêu chuẩn để gây nhiễm virus dịch tả lợn. Sau đây là hình ảnh tế bào PK15a sau 24 giờ nuôi cấy chụp qua kính hiển vi với độ phóng đại 10x20. Hầu hết những nghiên cứu về virus dịch tả lợn trên thế giới đều sử dụng các dòng tế bào có nguồn gốc từ dòng tế bào thận lợn, khả năng tăng trưởng của virus dịch tả lợn mạnh bắt đầu từ 48 giờ sau gây nhiễm và đạt cao nhất là 120 giờ trên tế bào PK15 (Leifer và ctv, 2010).
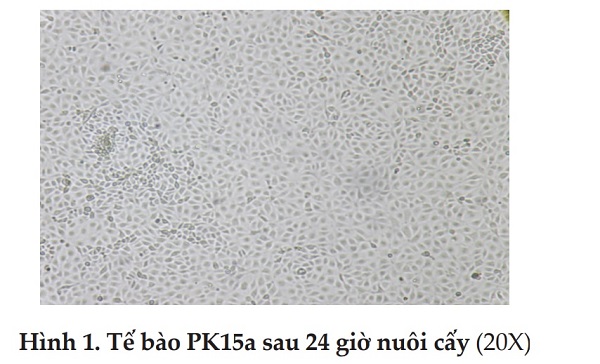
3.2. Kết quả gây nhiễm virus dịch tả lợn trên dòng tế bào PK15a
Sau khi có tế bào PK15a phát triển tốt, phủ đầy đáy chai 100%, tiến hành gây nhiễm virus dịch tả lợn chủng từ thực địa theo quy trình hướng dẫn của OIE (Hình 2) cho thấy sau 4 ngày theo dõi tế bào PK15a gây nhiễm virus dịch tả lợn các tế bào PK15a vẫn bám đáy rất tốt, vẫn giữ được hình thái đặc trưng của tế bào và không có bệnh tích tế bào gây ra bởi virus dịch tả lợn. Tiến hành thu tế bào để tách chiết RNA tổng số kiểm tra bằng phản ứng RT-PCR để kiểm tra sự có mặt của virus dịch tả lợn sau gây nhiễm.

Hình 2. Tế bào PK15a gây nhiễm virus dịch tả lợn với độ phóng đại 40X
3.3. Tối ưu hóa điều kiện phản ứng RT-PCR
Để xác định điều kiện tối ưu về nhiệt độ bắt cặp của cặp primer chẩn đoán vùng noncoding E2 của virus dịch tả lợn, sử dụng khuôn cDNA chuẩn của virus dịch tả lợn. Kết quả được chạy điện di trên thạch agarose 2%, 100V trong 25 phút được thể hiện trên hình 3.
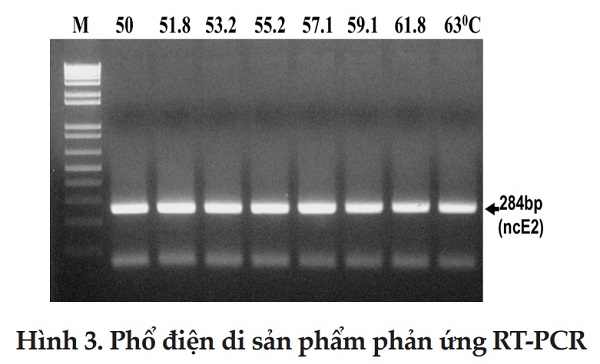
M: thang ADN chuẩn 1kb; giếng 1-8: sản phẩm RT-PCR Kết quả của phản ứng PCR gradient kiểm tra nhiệt độ bắt cặp tối ưu của mồi (Hình 3) cho thấy xuất hiện một băng ADN kích thước khoảng dưới 300bp đối với cả 8 nhiệt độ bắt cặp khác nhau 50-63°C. Kích thước này phù hợp với kích thước dự kiến của gen ncE2 của virus dịch tả lợn theo thiết kế khi được khuếch đại với cặp mồi đặc hiệu (284bp). Từ kết quả hình 3 cho thấy cặp mồi CSF324/326 cho hoạt động tốt và không có hiện tượng dimer, nhiệt độ bắt cặp tối ưu cho phản ứng PCR khuếch đại vùng noncoding gen E2 của virus dịch tả lợn là 50-63°C. Một số nghiên cứu giải mã trình tự gen và kỹ thuật phát hiện virus dịch tả lợn bằng kỹ thuật RT-PCR cũng cho thấy nhiệt độ bắt mồi gen E2 khoảng 50-63°C là nhiệt độ tối ưu phù hợp cho kết quả tốt (Rios và ctv, 2017; Elina và ctv, 2017).
3.4. Ứng dụng RT-PCR phát hiện đoạn gen noncoding E2 Virus dịch tả lợn
Từ kết quả phân lập virus dịch tả lợn từ thực địa tại tỉnh Bắc Giang, tiến hành tách chiết RNA và tổng hợp cDNA theo phương pháp mô tả tại phần phương pháp nghiên cứu. Phản ứng PCR được thực hiện dựa trên kết quả đã tối ưu tại phần 3.3. Kết quả phản ứng RT-PCR phát hiện đoạn gen ncE2 của virus dịch tả lợn (Hình 4).
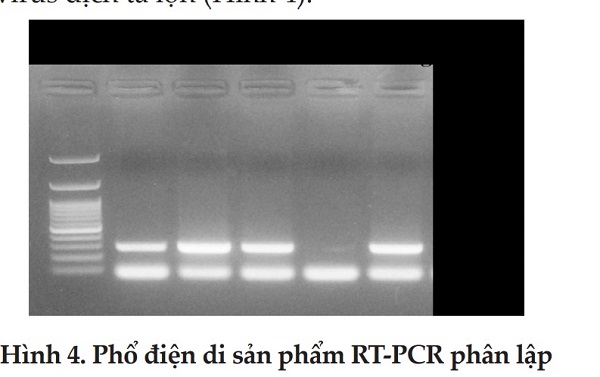
Kết quả kiểm tra RT-PCR sau gây nhiễm virus dịch tả lợn trên tế bào PK15a thể hiện qua hình 4 cho thấy sau 48h gây nhiễm virus dịch tả lợn trên tế bào PK15a đã cho kết quả dương tính với phản ứng RT-PCR. Như vậy, cặp mồi CSF324/326 khuếch đại vùng ncE2 của virus dịch tả lợn được thiết kế cho kết quả đặc hiệu với chủng virus dịch tả lợn phân lập từ thực địa.
Trong những năm gần đây, các kỹ thuật chẩn đoán nhanh bệnh dịch tả lợn đã được công bố. Vishal Chander và ctv (2014) nghiên cứu phương pháp chẩn đoán nhanh dịch tả lợn bằng phương pháp miễn dịch học, kết hợp với biểu hiện lâm sàng. Hoffmann và ctv (2005) nghiên cứu phát triển kỹ thuật RT-PCR để chẩn đoán nhanh và chính xác bệnh dịch tả lợn. Với kỹ thuật hiện đại, cho phép phát hiện sớm virus dịch tả lợn từ dịch họng của lợn mà không cần lấy máu (Stefano và ctv, 2018). Tuy nhiên, các kỹ thuật chẩn đoán bằng sinh học phân tử dựa trên những gen quan trọng như E2, 5’-NTR và NS5B cho kết quả nhanh và chính xác hơn (Sandra và ctv, 2017; Elina và ctv, 2017).
3.5. Xác định trình tự nucleotide của đoạn gen ncE2 virus dịch tả lợn
Trình tự gen và khung đọc của đoạn gen ncE2 của virus dịch tả lợn đã được tách dòng thể hiện trên hình 5 cho thấy trình tự đoạn gen ncE2 của virus dịch tả lợn được sử dụng để blast lên ngân hàng dữ liệu NCBI. Kết quả cho thấy đoạn gen ncE2 của virus dịch tả lợn ở nghiên cứu này có độ tương đồng 99% so với gen ncE2 của virus dịch tả lợn đã công bố. Khi so sánh trình tự nhận thấy gen ncE2 được tách dòng chỉ có 1 nucleotide khác so với dòng đã công bố và trình tự amino axit là tương đồng tại điểm sai khác.
Từ kết quả trên cho thấy cặp primer CSF324/326 được thiết kế cho kết quả tương đồng và có độ đặc hiệu với chủng phân lập tại thực địa.
4. KẾT LUẬN
Đã phân lập được virus dịch tả lợn chủng thực địa trên dòng tế bào PK15a Cặp mồi CSF324/326 có thể dùng để chẩn đoán virus dịch tả lợn lưu hành tại Việt Nam. Có thể sử dụng cặp mồi CSF324/326 dùng để phân tích di truyền virus dịch tả lợn lưu hành tại Việt Nam.
TÀI LIỆU THAM KHẢO
1. Bhaskar N., Ravishankar C., Rajasekhar R., Sumod K., Sumithra T.G and John K. (2015). Molecular typing and phylogenetic analysis of classical swine fever virus isolates from Kerala, India. Virus disease, 26(4): 260-66.
2. Nguyễn Tiến Dũng (1999). Dịch tả lợn cổ điển luôn là vấn đề thời sự. Tạp chí KHKT Thú y, 4(2): 48-55. 3. Nguyễn Thị Phương Duyên, Đỗ Văn Khuyên và Dư Đình Quân (2001). Khảo sát hội chứng sốt, táo bón bỏ ăn đến chết ở đàn lợn sinh sản và đàn lợn con tỉnh Khánh Hòa và Quảng Ngãi. Báo cáo KHCNTY, Bộ NN và PTNT, 10-12/4/2001.
4. Đào Trọng Đạt và Nguyễn Tiến Dũng (1985). Về tình hình dịch tễ của bệnh dịch tả lợn cổ điển ở Việt nam và vấn đề phòng chống bệnh. Tuyển tập các công trình nghiên cứu khoa học và kỹ thuật nông nghiệp (1981-1985)-Phần Chăn nuôi -Thú y.
5. Elina K., Nagendra N.B., Manab D., Gitika R., Kongkon B., Nipu D., Durlav P.B. and Sachin K. (2017). Molecular characterization of classical swine fever virus isolates from India during 2012-14. Acta Tropica, 170: 180-89.
6. Hoffmann B., Beer M., Schelp C., Schirrmeier H., and Depner K. (2005). Validation of a real-time RT-PCR assay for sensitive and specific detection of classical swine fever. J. Virol Methods, 130(1-2): 36-44.
7. Leifer I., Everett H., Hoffmann B., Sosan O., Crooke H. and Beer M. (2010). Escape of classical swine fever C-strain vaccine virus from detection by C-strain specific real-time RT-PCR caused by a point mutation in the primer-binding site. J. Virol Methods, 166(1-2): 98-00.
8. Rios L., Coronado L., Naranjo-Feliciano D., Martinez[1]Perez O., Perera C.L. and Hernandez-Alvarez L. (2017). Deciphering the emergence, genetic diversity and evolution of classical swine fever virus. Sci. Rep., 7(1): 17887. 9. Sandra B., Christoph S., Julia H., Jolene C. and Martin B. (2017). Classical swine fever-an updated review. Viruses J., 86: 1-24. 10. Stefano P., Ilaria P., Monica G., Francesco F. and Gian M.D.M. (2018). Detection of Classical swine fever virus infection by individual oral fluid of pigs following experimental inoculation. J. Vet. Dia. Inv., 29(2): 254-257. 11. Vishal C., S. Nandi, C. Ravishankar, V. Upmanyu and Rishendra V. (2014). Classical swine fever in pigs: recent developments and future perspectives. Anim. Hea. Res. Rev., 15(1): 87-01.